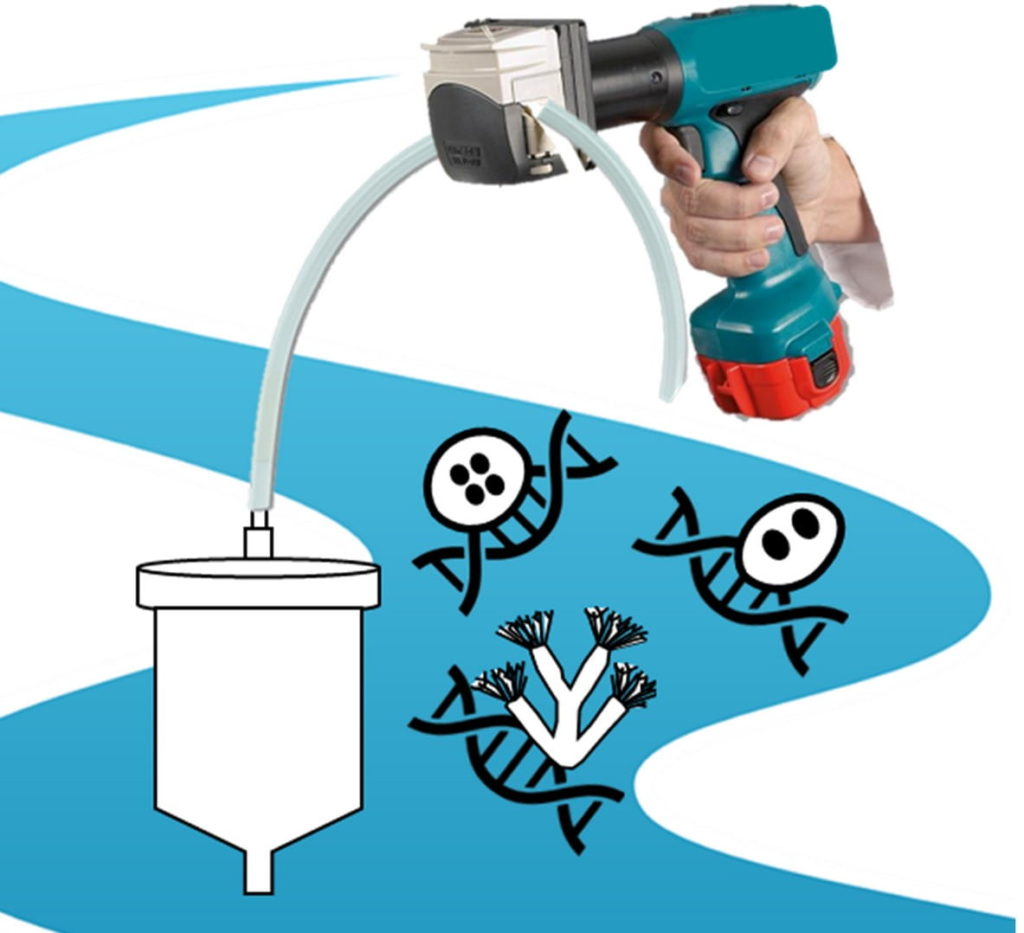
La MRP (ou PKD pour proliferative kidney disease) est une maladie infectieuse émergente causée par le parasite Tetracapsuloides bryosalmonae. Au cours des deux dernières décennies, de plus en plus d’épidémies de MRP ont été reportées au sein de populations de salmonidés d’Europe et d’Amérique du Nord (Hedrick et al., 1993; Sudhagar et al., 2020). Cette maladie est en effet responsable de pertes importantes en aquaculture et de déclins inquiétants des populations de salmonidés sauvages, avec jusqu’à 95 % de mortalité signalée dans les fermes piscicoles (Okamura et al., 2011). En outre, plusieurs études ont montré que le déclin de la truite fario (Salmo trutta) dans les rivières suisses était lié au développement de la MRP (Burkhardt-Holm & Scheurer, 2007; Wahli et al., 2007). En France, la distribution du parasite et le statut d’infection des populations de salmonidés sauvages restent encore largement méconnus.
T. bryosalmonae est un parasite myxozoaire qui alterne entre deux hôtes au cours de son cycle de vie: un hôte bryozoaire et un hôte poisson salmonidé (Fig. 1). Les bryozoaires sont des animaux coloniaux ressemblant à de la mousse qui vivent attachés au substrat, souvent dans de petites cavités, aux racines des arbres de la ripisylve, ou encore dans des habitats artificiels submergés (Hartikainen & Okamura, 2015; T. S. Wood & Marsh, 1999). Lorsque les conditions environnementales sont favorables au développement des bryozoaires (augmentation de la température de l’eau et de l’abondance des nutriments), la libération des spores de T. bryosalmonae par les colonies infectées débute (Fontes et al., 2017; Gay et al., 2001). Les spores parasites pénètrent ensuite dans leur hôte poisson par les branchies ou la peau, circulent dans le sang et se multiplient lorsqu’elles atteignent leurs organes cibles, principalement les reins et la rate (Okamura et al., 2011). L’infection peut rester bénigne, dans ce cas les poissons infectés sont des porteurs sains. Cependant, la prolifération des parasites dans le tissu interstitiel rénal peut provoquer une réponse immunitaire excessive, responsable d’un gonflement des reins (Chilmonczyk et al., 2002). Cela entraîne une altération des fonctions rénales, y compris de la production de cellules sanguines, avec un risque d’anémie et une diminution du transport de l’oxygène pouvant éventuellement entraîner la mort des poissons par asphyxie (Hedrick et al., 1993; Palikova et al., 2017). La limite entre l’infection bénigne et le développement de la MRP est étroitement liée à la température de l’eau, et les symptômes de la maladie sont principalement signalés lorsque celle-ci atteint 15°C (Bailey et al., 2017; Clifton-Hadley et al., 1986). Les poissons qui survivent à leur première infection par T. bryosalmonae développent vraisemblablement une réponse immunitaire adaptative car ils ne développent pas la MRP lors de réinfection ultérieure (Feist & Longshaw, 2006; Foott & Hedrick, 1987). Ainsi, les jeunes de l’année, par définition exposés pour la première fois, sont les plus concernés par les conséquences de la maladie.

Figure 1: Cycle de vie de T. bryosalmonae et champ d’action des méthodes ADNu et ADNe (description ci-dessous). Les spores libérées par les poissons ne sont infectieuses que pour les bryozoaires et vice-versa : il n’y a pas de contamination de poisson à poisson.
Après multiplication dans l’interstitium rénal des poissons, les spores migrent dans la lumière des tubules rénaux (Morris & Adams, 2008) et sont excrétées dans l’eau de la rivière via les urines du poisson infecté (Hedrick et al., 2004). Ces spores sont infectieuses uniquement pour les hôtes bryozoaires. Jusqu’à présent, seule la transmission depuis la truite fario et l’omble de fontaine (Salvelinus fontinalis) de T. bryosalmonae aux colonies de bryozoaires a pu être prouvée (Grabner & El-Matbouli, 2008), suggérant que les autres poissons hôtes pourraient représenter des impasses pour le parasite.
L’identification des zones à risque pour le développement de la MRP nécessite d’attester (i) la présence du parasite et de son hôte bryozoaire dans l’environnement, (ii) le statut d’infection des poissons.
L’ADN environnemental (ADNe) désigne l’ADN libéré par les organismes qui peut ensuite se retrouver dans les échantillons d’eau, de sol ou de fèces, témoignant de la présence d’une espèce (Bohmann et al., 2014; Pawlowski et al., 2020). La détection d’ADNe est rapidement devenue un standard dans les études écologiques en raison de la disponibilité croissante d’outils moléculaires puissants permettant la détection d’espèces même à partir d’une faible quantité d’ADN disponible dans l’environnement (Jerde et al., 2011; Miotke et al., 2014; Shokralla et al., 2012). Dans le cas de maladies infectieuses, l’ADNe peut être utilisé pour détecter des parasites provenant soit de l’environnement ouvert (c’est-à-dire l’air, l’eau, le sol) pour obtenir des informations sur l’exposition de l’hôte (Rusch et al., 2018), soit des fluides de l’hôte (le sang, les fèces ou l’urine), ce qui permet d’obtenir le statut d’infection de l’hôte individuel, ainsi qu’un suivi individuel tout au long des stades de l’infection (Etienne et al., 2012).
Dans ce cadre, SCIMABIO-Interface et Fish-Pass proposent l’utilisation d’outils opérationnels basés sur la détection d’ADN environnemental, et adaptés de précédentes recherches menées dans le cadre d’une thèse (Duval, 2022; Duval et al., 2021).
(i) Détection du parasite et de son hôte bryozoaire dans les eaux de rivière (ADNe)
Cette méthode consiste en la filtration d’un échantillon d’eau de rivière, et à la recherche ciblée de l’ADN du parasite T. bryosalmonae et de son hôte bryozoaire Fredericella sultana (Carraro et al., 2018).
Après extraction de l’ADN, des amorces spécifiques à ces deux espèces sont utilisées pour amplifier simultanément leur ADN. La méthode utilisée est la droplet digital Polymerase Chain Reaction (ddPCR, Fig. 3). La ddPCR permet de détecter de l’ADN présent en infime quantité dans un échantillon, ce qui la rend plus sensible que les PCRs classiques (Doi et al., 2015; S. A. Wood et al., 2019).
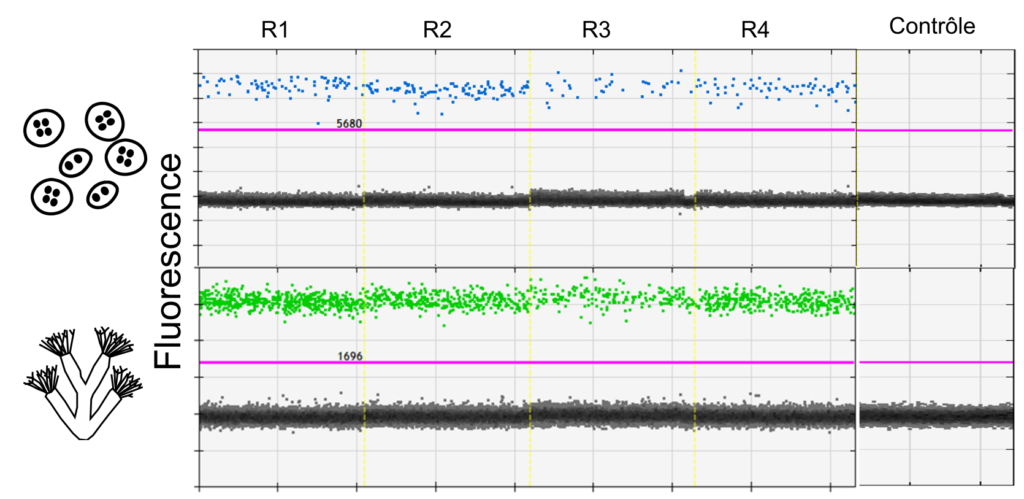
Figure 3: Visualisation des résultats de la ddPCR pour un site avec ici 4 réplicas terrain (R1, R2, R3, R4). Les gouttelettes positives dans lesquelles de l’ADN est présent fluorescent en bleu pour T. bryosalmonae et en vert pour F. sultana. Un contrôle négatif est également représenté, et la ligne rose indique le seuil au-dessus duquel les gouttelettes sont considérées comme positives. Il est déterminé à partir d’un témoin positif. ©E. Duval
En utilisant l’ADNe, il est donc possible de cartographier la présence/absence des deux espèces cibles (parasite et bryozoaire) sur une large zone géographique (ex. Fig. 4), tout en estimant leur abondance relative. Cette méthode facilement déployable sur le terrain permet également d’identifier de façon non invasive (pas de capture d’individus) les zones prioritaires pour un suivi d’infection chez les poissons. De cette façon, des études spatiales et/ou temporelles peuvent être mises en place.
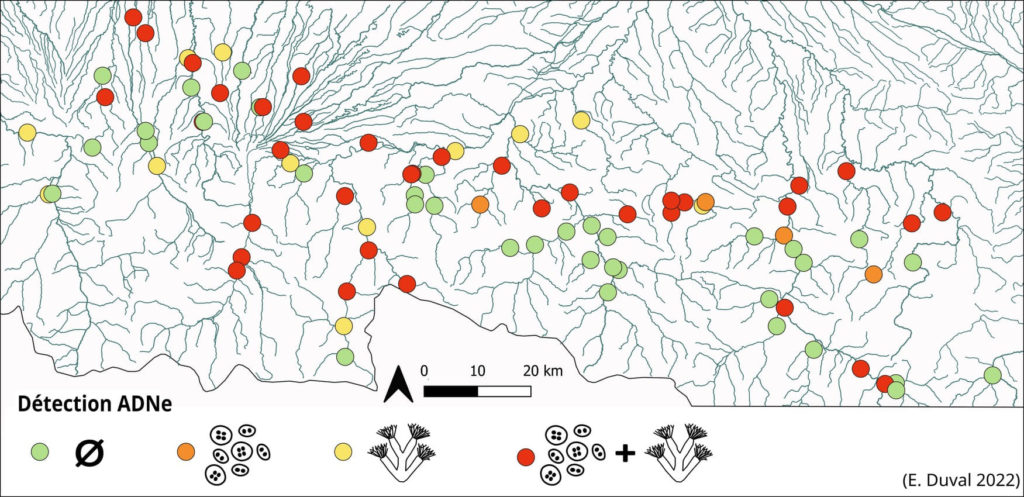
Figure 4: Cartographie de la présence/absence de T. bryosalmonae et F. sultana sur 87 sites d’études répartis à l’échelle d’un bassin versant.
(ii) Diagnostic du statut d’infection des individus de truite fario (ADNu)
Cette fois-ci, l’ADN du parasite n’est pas recherché dans le milieu (eau de rivière), mais dans les excrétions des poissons testés en se basant sur le cycle de vie du parasite (Fig. 1, Fig. 5). Cette méthode, baptisée ADNu pour ADN urinaire (Duval et al., 2021), permet grâce à l’amplification par ddPCR (voir ci-dessus) de l’ADN du parasite de discriminer les individus infectés par T. bryosalmonae des individus non infectés, et d’ainsi estimer une prévalence d’infection au sein des populations testées (Fig. 6).

Figure 5 : Déroulement des opérations sur le terrain depuis la pêche des individus, la maintenance des individus testés dans une eau non contaminée et la filtration de cette eau à partir de laquelle l’extraction d’ADN pourra être réalisée.
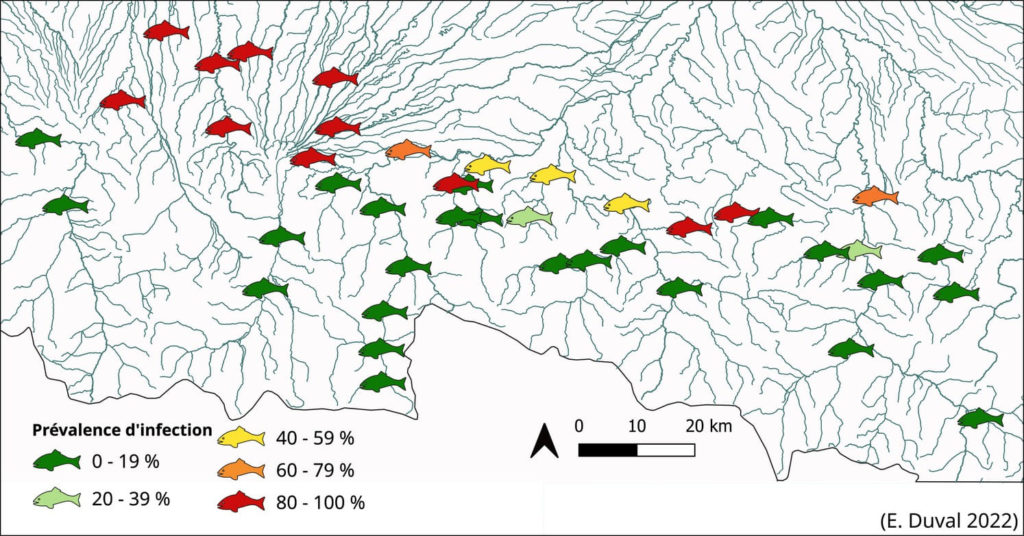
Figure 6 : Exemple d’application et cartographie de la prévalence d’infection de populations réparties à l’échelle d’un bassin versant.
Cette méthode ADNu est jusqu’à aujourd’hui la seule qui permette de déceler l’infection de façon non létale, les autres méthodes requérant un prélèvement de rein. Aussi, l’utilisation de l’ADNu ne permet pas d’attester du potentiel développement de la maladie (présence de symptômes), seulement de l’infection.
Que ce soit pour une étude ADNu ou ADNe, SCIMABIO-Interface et Fish-Pass gèrent le suivi de l’étude de A à Z depuis les conseils pour la stratégie d’échantillonnage, en passant par la formation aux manipulations sur le terrain, aux analyses en laboratoire et à l’interprétation écologique des résultats. Une interprétation plus poussée peut être envisagée dans le cadre d’un couplage avec des données environnementales disponibles telles que la qualité de l’eau ou la température.
Références citées :
Bailey, C., Segner, H., Casanova-Nakayama, A., & Wahli, T. (2017). Who needs the hotspot? The effect of temperature on the fish host immune response to Tetracapsuloides bryosalmonae the causative agent of proliferative kidney disease. Fish & Shellfish Immunology, 63, 424‑437. https://doi.org/10.1016/j.fsi.2017.02.039
Bohmann, K., Evans, A., Gilbert, M. T. P., Carvalho, G. R., Creer, S., Knapp, M., Yu, D. W., & de Bruyn, M. (2014). Environmental DNA for wildlife biology and biodiversity monitoring. Trends in Ecology & Evolution, 29(6), Article 6. https://doi.org/10.1016/j.tree.2014.04.003
Burkhardt-Holm, P., & Scheurer, K. (2007). Application of the weight-of-evidence approach to assess the decline of brown trout (Salmo trutta) in Swiss rivers. Aquatic Sciences, 69(1), Article 1. https://doi.org/10.1007/s00027-006-0841-6
Carraro, L., Hartikainen, H., Jokela, J., Bertuzzo, E., & Rinaldo, A. (2018). Estimating species distribution and abundance in river networks using environmental DNA. Proceedings of the National Academy of Sciences, 201813843. https://doi.org/10.1073/pnas.1813843115
Chilmonczyk, S., Monge, D., & de Kinkelin, P. (2002). Proliferative kidney disease : Cellular aspects of the rainbow trout, Oncorhynchus mykiss (Walbaum), response to parasitic infection. Journal of Fish Diseases, 25(4), Article 4. https://doi.org/10.1046/j.1365-2761.2002.00362.x
Clifton-Hadley, R. S., Richards, R. H., & Bucke, D. (1986). Proliferative kidney disease (PKD) in rainbow trout Salmo gairdneri : Further observations on the effects of water temperature. Aquaculture, 55(3), Article 3. https://doi.org/10.1016/0044-8486(86)90112-2
Doi, H., Takahara, T., Minamoto, T., Matsuhashi, S., Uchii, K., & Yamanaka, H. (2015). Droplet Digital Polymerase Chain Reaction (PCR) Outperforms Real-Time PCR in the Detection of Environmental DNA from an Invasive Fish Species. Environmental Science & Technology, 49(9), Article 9. https://doi.org/10.1021/acs.est.5b00253
Duval, E. (2022). Detection, distribution, and impacts of the emerging parasite Tetracapsuloides bryosalmonae on wild populations of the brown trout Salmo trutta [Thèse de doctorat, 239p]. Université Toulouse III Paul Sabatier.
Duval, E., Blanchet, S., Quéméré, E., Jacquin, L., Veyssière, C., Lautraite, A., Garmendia, L., Yotte, A., Parthuisot, N., Côte, J., & Loot, G. (2021). Urine DNA (uDNA) as a non‐lethal method for endoparasite biomonitoring : Development and validation. Environmental DNA, edn3.228. https://doi.org/10.1002/edn3.228
Etienne, L., Locatelli, S., Ayouba, A., Esteban, A., Butel, C., Liegeois, F., Aghokeng, A., Delaporte, E., Mpoudi Ngole, E., & Peeters, M. (2012). Noninvasive Follow-Up of Simian Immunodeficiency Virus Infection in Wild-Living Nonhabituated Western Lowland Gorillas in Cameroon. Journal of Virology, 86(18), Article 18. https://doi.org/10.1128/JVI.01186-12
Feist, S. W., & Longshaw, M. (2006). Phylum Myxozoa. In P. T. K. Woo, Fish diseases and disorders (CAB International, p. 230‑296).
Fontes, I., Hartikainen, H., Williams, C., & Okamura, B. (2017). Persistence, impacts and environmental drivers of covert infections in invertebrate hosts. Parasites & Vectors, 10(1), Article 1. https://doi.org/10.1186/s13071-017-2495-8
Foott, J. S., & Hedrick, R. P. (1987). Seasonal occurrence of the infectious stage of proliferative kidney disease (PKD) and resistance of rainbow trout, Salmo gairdneri Richardson, to reinfection. Journal of Fish Biology, 30(4), Article 4. https://doi.org/10.1111/j.1095-8649.1987.tb05771.x
Gay, M., Okamura, B., & de Kinkelin, P. (2001). Evidence that infectious stages of Tetracapsula bryosalmonae for rainbow trout Oncorhynchus mykiss are present throughout the year. Diseases of Aquatic Organisms, 46, 31‑40. https://doi.org/10.3354/dao046031
Grabner, D., & El-Matbouli, M. (2008). Transmission of Tetracapsuloides bryosalmonae (Myxozoa : Malacosporea) to Fredericella sultana (Bryozoa : Phylactolaemata) by various fish species. Diseases of Aquatic Organisms, 79, 133‑139. https://doi.org/10.3354/dao01894
Hartikainen, H., & Okamura, B. (2015). Ecology and Evolution of Malacosporean-Bryozoan Interactions. In B. Okamura, A. Gruhl, & J. L. Bartholomew, Myxozoan Evolution, Ecology and Development (Springer International Publishing, p. 201‑216).
Hedrick, R. P., Baxa, D. V., De Kinkelin, P., & Okamura, B. (2004). Malacosporean-like spores in urine of rainbow trout react with antibody and DNA probes to Tetracapsuloides bryosalmonae. Parasitology Research, 92(1), Article 1. https://doi.org/10.1007/s00436-003-0986-3
Hedrick, R. P., MacConnell, E., & de Kinkelin, P. (1993). Proliferative kidney disease of salmonid fish. Annual Review of Fish Diseases, 3, 277‑290. https://doi.org/10.1016/0959-8030(93)90039-E
Jerde, C. L., Mahon, A. R., Chadderton, W. L., & Lodge, D. M. (2011). “Sight-unseen” detection of rare aquatic species using environmental DNA. Conservation Letters, 4(2), Article 2. https://doi.org/10.1111/j.1755-263X.2010.00158.x
Miotke, L., Lau, B. T., Rumma, R. T., & Ji, H. P. (2014). High Sensitivity Detection and Quantitation of DNA Copy Number and Single Nucleotide Variants with Single Color Droplet Digital PCR. Analytical Chemistry, 86(5), Article 5. https://doi.org/10.1021/ac403843j
Morris, D. J., & Adams, A. (2008). Sporogony of Tetracapsuloides bryosalmonae in the brown trout Salmo trutta and the role of the tertiary cell during the vertebrate phase of myxozoan life cycles. Parasitology, 135(09), Article 09. https://doi.org/10.1017/S0031182008004605
Okamura, B., Hartikainen, H., Schmidt-Posthaus, H., & Wahli, T. (2011). Life cycle complexity, environmental change and the emerging status of salmonid proliferative kidney disease. Freshwater Biology, 56(4), Article 4. https://doi.org/10.1111/j.1365-2427.2010.02465.x
Palikova, M., Papezikova, I., Markova, Z., Navratil, S., Mares, J., Mares, L., Vojtek, L., Hyrsl, P., Jelinkova, E., & Schmidt-Posthaus, H. (2017). Proliferative kidney disease in rainbow trout (Oncorhynchus mykiss) under intensive breeding conditions : Pathogenesis and haematological and immune parameters. Veterinary Parasitology, 238, 5‑16. https://doi.org/10.1016/j.vetpar.2017.03.003
Pawlowski, J., Apothéloz‐Perret‐Gentil, L., & Altermatt, F. (2020). Environmental DNA : What’s behind the term? Clarifying the terminology and recommendations for its future use in biomonitoring. Molecular Ecology, 29(22), Article 22. https://doi.org/10.1111/mec.15643
Rusch, J. C., Hansen, H., Strand, D. A., Markussen, T., Hytterød, S., & Vrålstad, T. (2018). Catching the fish with the worm : A case study on eDNA detection of the monogenean parasite Gyrodactylus salaris and two of its hosts, Atlantic salmon (Salmo salar) and rainbow trout (Oncorhynchus mykiss). Parasites & Vectors, 11(1), Article 1. https://doi.org/10.1186/s13071-018-2916-3
Shokralla, S., Spall, J. L., Gibson, J. F., & Hajibabaei, M. (2012). Next-generation sequencing technologies for environmental DNA research. Molecular Ecology, 21(8), Article 8. https://doi.org/10.1111/j.1365-294X.2012.05538.x
Sudhagar, A., Kumar, G., & El-Matbouli, M. (2020). The Malacosporean Myxozoan Parasite Tetracapsuloides bryosalmonae : A Threat to Wild Salmonids. Pathogens, 9(1), Article 1. https://doi.org/10.3390/pathogens9010016
Wahli, T., Bernet, D., Steiner, P. A., & Schmidt-Posthaus, H. (2007). Geographic distribution of Tetracapsuloides bryosalmonae infected fish in Swiss rivers : An update. Aquatic Sciences, 69(1), Article 1. https://doi.org/10.1007/s00027-006-0843-4
Wood, S. A., Pochon, X., Laroche, O., Ammon, U., Adamson, J., & Zaiko, A. (2019). A comparison of droplet digital polymerase chain reaction (PCR), quantitative PCR and metabarcoding for species‐specific detection in environmental DNA. Molecular Ecology Resources, 19(6), Article 6. https://doi.org/10.1111/1755-0998.13055
Wood, T. S., & Marsh, T. G. (1999). Biofouling of wastewater treatment plants by the freshwater bryozoan, Plumatella vaihiriae (Hastings, 1929). Water Research, 33(3), Article 3. https://doi.org/10.1016/S0043-1354(98)00274-7