Projet terminé (2022-2023)

Contexte et objectifs
La moule perlière d’eau douce (Margaritifera margaritifera, Figure 1) est une espèce patrimoniale en déclin dans les cours d’eau de France et d’Europe en raison de la dégradation de la qualité de l’eau et de son habitat, combinée aux effets du changement climatique. Si son statut de menace en Europe est « en danger critique », son statut en France est « en danger » puisqu’elle est recensée dans la Liste rouge des mollusques continentaux de France métropolitaine (IUCN, 2021).
Plusieurs populations de moules perlières sont connues et suivies en France métropolitaine, notamment dans les rivières du Massif central (Puissauve et al. 2015). Plusieurs rivières de Haute-Loire (43) sont connues pour avoir abritées historiquement l’espèce, certaines populations étant clairement identifiées et suivies comme sur le Lignon au niveau du Pont de Mars. Cependant les populations tendent à décliner progressivement avec le temps et la mise en place des suivis traditionnels en prospection visuelle difficile à mettre en place sur l’ensemble des rivières du département.
L’objectif de cette étude est donc d’utiliser des approches basées sur l’ADN environnemental (ADNe) pour évaluer la présence de la moule perlière au niveau de 19 stations localisées sur 10 rivières de département de la Haute-Loire. Ce diagnostic de présence a pour finalité d’accompagner l’Epage Loire-Lignon dans l’acquisition de connaissance quant à la distribution de l’espèce pour prioriser des secteurs sur lesquels déployer des suivis traditionnels et des actions en faveur de la conservation de l’espèce.
Méthodologie
Le choix des stations a été effectué en concertation avec l’Epage Loire-Lignon en fonction des données historiques de présence de la moule perlière sur les cours d’eau de Haute-Loire ; les habitats et conditions en théories favorables à la présence de l’espèce ; l’accessibilité des sites. Au total 19 stations réparties sur 10 rivières du département 43 ont été retenues (Figure 2). Avec 2 prélèvements par station, un total de 38 prélèvements d’eau a été collecté par Scimabio avec l’appui technique de l’Epage.
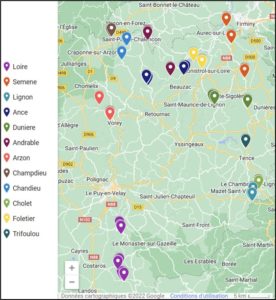
Figure 2 : Emplacement des 19 stations et 38 points de prélèvement d’eau réalisés sur les 10 rivières de l’étude
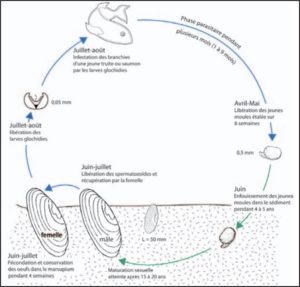
Figure 3 : Cycle de reproduction théorique de Margaritifera margaritifera en Europe (Cochet & Paris 2005)
En période de reproduction (Juin-Juillet), les individus mâles relarguent des gamètes dans l’eau qui sont récupérés par les individus femelles par filtration (Cochet & Paris 2005). Après fécondation des ovules, les cellules œufs incubent pendant environ 4 semaines dans les individus femelles avant d’être relarguées sous forme de larves appelées glochidies. Chaque femelle peut ainsi relarguer plusieurs centaines de milliers de larves qui vont continuer le cycle de vie de l’espèce en parasitant les branchies de poissons, principalement des salmonidés (Figure 3).
Afin de maximiser les chances de détection de l’espèce par ADNe, les prélèvements d’eau ont donc été réalisés en Août 2022. Cette période coïncide en théorie avec le chevauchement du relargage des gamètes par les individus mâles et des larves glochidies par les individus femelles dans l’eau (Wacker et al. 2019).
Les échantillonnages ont été réalisés pour chaque station de la manière suivante :
- 4 prélèvements de 500mL d’eau collectés à environ 5-20 cm de profondeur sur des zones de radiers ou en sortie de courant, ce de manière à couvrir la largeur du cours d’eau. Les 4 échantillons ont ensuite été « poolés » en un échantillon unique de 2L conservé au frais en attendant la filtration.
- Cette stratégie d’échantillonnage a été réalisée 2 fois par station afin d’avoir 2 réplicats de terrain. Dans la mesure du possible les 2 réplicats de terrain nommés A et B doivent être distants d’au moins 50-100m pour prendre en compte la variabilité locale du signal.
Les extractions ont été faites avec le kit d’extraction « NucleoMag DNA/RNA Water kit » (Macherey-Nagel) en suivant les recommandations d’extractions à partir de filtre Sterivex. La qualité et la quantité d’ADN total extrait pour chaque prélèvement ont été mesurées au spectrophotomètre NanoDrop™ One/OneC (ThermoFisher). Les dPCR ont été réalisées grâce au Naica® system for Crystal Digital PCR™ (Stilla). Des témoins négatifs d’extractions et de dPCR ont été intégrés aux analyses afin d’évaluer des contaminations potentielles introduites par l’opérateur ainsi que la présence de contaminations croisées entre échantillons. Afin de comparer le signal ADNe de la moule perlière avec celui d’une espèce repère environnementale, une démarche similaire a été entreprise avec le Vairon. 2 signaux ont donc été recherchés dans les échantillons ADNe : le signal cible Moule perlière mitochondrial et le signal Vairon mitochondrial servant de contrôle positif.
Principaux résultats
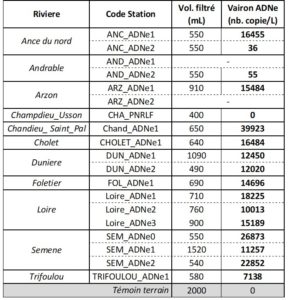
Table 1 : Signal ADNe Vairon détecté par dPCR sur les 19 stations fléchées pour la détection de la moule perlière.
Un signal ADNe vairon positif a pu être identifié sur l’ensemble des 19 stations étudiées pour la recherche de la moule perlière à l’exception de la station sur le Champdieu au niveau d’Usson-en-Forez (Table 1). Le nombre de copies d’ADN mitochondrial de Vairon retrouvé par Litre d’eau filtré varie d’une station à une autre, allant de 36 à 39923 copies/Litre (stations ANC_ADNe2 et Chand_ADNe1 respectivement). Si la majorité des détections est entre 10000 et 40000 copies/Litre, il est important de noter que les détections sur les stations ANC_ADNe2 (36 copies/L) et AND_ADNe2 (55 copies/L) sont très faibles au regard des autres stations.
Ces résultats étaient attendus car l’espèce est présente de manière assez abondante sur une grande majorité des cours d’eau du département de la Haute-Loire. Il est important de noter que cette détection a été faite en pleine période de reproduction et d’émergence de l’espèce. Le signal ADNe détecté est donc à son niveau « maximal » car il correspond à l’ADN libéré par les individus et au signal associé à son cycle de vie (déplacement, reproduction, émergence…). C’est pour cette raison que les signaux ADNe détectés pour le vairon sont particulièrement élevés avec plusieurs milliers de copies par Litre.
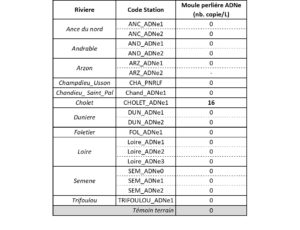
Sur les 19 stations de l’étude dédiée à la recherche de présence de la moule perlière, une seule station présente un signal ADNe positif correspondant à la station CHOLET_ADNe1 sur le Cholet (Table 2). Le nombre de copies d’ADN mitochondrial de moule perlière retrouvé sur cette station est en moyenne 16 copies/Litre d’eau. Cette valeur est assez faible au regard des signaux détectés sur les 4 stations du Pont de Mars.
Aucun signal ADNe moule perlière n’a été détecté dans le témoin négatif de terrain, confirmant l’absence de contamination lors de l’analyse.
Références bibliographiques
- Site internet de l’IUCN pour la liste rouge des espèces à enjeux de protection : https://www.iucnredlist.org/species/12799/212995510
- Cochet, G., & Paris, L., 2005. La moule perlière Margaritifera margaritifera (Linnaeus, 1758) et son statut dans le Morvan. Rev. sci. Bourgogne-Nature 2-2005, 19-29.
- Wacker, S., Fossøy, F., Larsen, B. M., Brandsegg, H., Sivertsgård, R., & Karlsson, S. (2019). Downstream transport and seasonal variation in freshwater pearl mussel “Margaritifera margaritifera” eDNA concentration. Environmental DNA, 1 (1), 64–73. https://doi.org/10.1002/edn3.10.
Partenaires du projet