Utilisation de l’ADN environnemental pour la détection de l’écrevisse à pattes blanches (Austropotamobius pallipes) et de l’agent responsable de la peste de l’écrevisse (Aphanomyces astaci) en cours d’eau
Projet en cours (2023-2025)
Partenaires
SCIMABIO Interface (porteur)
Office français pour la biodiversité (OFB)
Université de Poitiers – EBI/EES

Contexte
L’écrevisse à pattes blanches (Austropotamobius pallipes) est l’une des 3 espèces autochtones retrouvée dans les cours d’eau d’Europe de l’Ouest. Bien que pouvant occuper des habitats assez diversifiés, elle est présente principalement dans les petits cours d’eau en tête de bassin versant là où les pressions sont généralement les moins importantes.
Au cours de la dernière décennie, les populations d’écrevisse à pattes blanches ont fortement décliné, plusieurs facteurs pouvant être mis en avant pour expliquer ce phénomène :
- La dégradation des milieux aquatiques, à la fois en termes de réduction des habitats (dégradation des berges) que de la qualité de l’eau (états écologique et chimique) ;
- L’introduction d’espèces d’écrevisse non indigènes (allochtones) envahissantes, plus agressives et compétitives (habitat, ressources) que les espèces autochtones ;
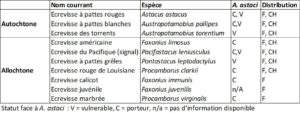
- L’apparition de maladies, souvent liée à l’introduction d’espèces allochtones, comme la peste de l’écrevisse causée par l’oomycète Aphanomyces astaci (Table 1).
De ce fait, l’écrevisse à pattes blanches est classée « en danger » sur la liste rouge mondiale de l’UICN depuis 2010 (Füreder et al. 2010) et comme “vulnérable” de manière générale en France métropolitaine (MNHN & OFB 2021). L’espèce fait l’objet de mesures règlementaires de protection (habitat, pêche, transport) et différents programmes de conservation sont mis en place par les gestionnaires.
Ces programmes nécessitent d’avoir des données de suivi robustes. Malheureusement, les données disponibles quant à la distribution de A. pallipes et des espèces affectant son écologie (écrevisses allochtones, agent de la peste A. astaci) sont souvent hétérogènes voire inexistantes dans certains cours d’eau de France métropolitaine.
Méthodologie
Le manque de données spatiales et temporelles réside dans la difficulté de mise en place des protocoles de détections des écrevisses (comptages nocturnes, piégeage) et de la peste (suivi d’écrevisses infectées, observation microscopique…). Leur application sur les cours d’eau requière des moyens humains et financiers importants, ce qui limite le nombre de suivis réalisables. De plus, les biais associés à certaines approches (échantillonnage, technique) donnent souvent une image incomplète de l’état des populations.
Au cours de la dernière décennie, des approches de biologie moléculaires utilisant l’ADN extrait à partir d’échantillons environnementaux (communément appelé ADNe) se sont fortement développées sur les milieux aquatiques. En fonction des matrices environnementales échantillonnées (eau, biofilm, sédiment) et des techniques déployées en laboratoire, il est possible de réaliser des inventaires de diversité ou le suivi d’espèces d’intérêt grâces à l’ADN des individus retrouvé dans l’environnement.
Plusieurs études ont déjà montré l’efficacité des approches moléculaires utilisant l’ADNe pour le suivi de différentes espèces d’écrevisses dont A. pallipes en cours d’eau ainsi que de la peste A. astaci (Chucholl et al. 2020, Troth et al. 2020, Baudry et al. 2021, King et al. 2022). Bien que des facteurs environnementaux et techniques rendent difficile le lien entre signal ADNe (nombre de copie du gène ciblé) et nombre d’individus (e.g. Dunn et al. 2017), les approches ADNe ont une bonne capacité de détection des espèces souvent supérieure aux approches traditionnelles (Figure 1).
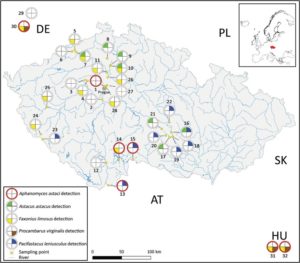
Figure 1 – Exemple de détection de plusieurs espèces d’écrevisses (A. astacus, F. limosus, P. virginalis, P. leniusculus) et de la peste (A.astaci) via des approches ADNe sur des cours d’eau en République tchèque (Rusch et al. 2020).
De plus de nouvelles pistes d’amélioration sont déployables pour rendre les approches ADNe plus robustes et accessibles à des suivis en routine :
- Caractérisation du signal ADNe en rivière : des études ont montré que les biofilms aquatiques sont des capteurs passifs de l’ADNe et ont le potentiel de mieux intégrer le signal ADNe par rapport à un prélèvement ponctuel d’eau (Rivera et al. 2020). Aux vues de l’écologie des écrevisses comme A. pallipes et de la peste A. astaci en cours d’eau, cette matrice semble bien adaptée à leur détection. L’ADNe extrait à partir de biofilms offre aussi la possibilité d’évaluer l’état écologique du cours d’eau via le suivi des communautés de diatomées (Vasselon et al. 2019), information importante lors de la mise en place de mesures de conservation (Manenti et al. 2021).
- Capacité de purification de l’ADNe : pour permettre la mise en place de la plupart des approches de biologie moléculaires sur l’ADNe, ce dernier doit être récupéré en quantité et qualité suffisante. Les méthodes couramment utilisées pour extraire l’ADN d’échantillons environnementaux reposent sur des approches de précipitations ou de purification sur colonne de silice. Si les méthodes de précipitations tendent à maximiser les quantités d’ADN obtenues au détriment de la qualité du fait de la présence de composées co-extrait (e.g. acides humiques), l’utilisation de colonne de purification augmentent quant à elle la qualité de l’ADN obtenu mais au détriment de la quantité. Dans les deux cas, l’utilisation de l’ADNe peut en être affecté au point de limiter la détection d’évènement rare voir d’empêcher tout travail en laboratoire (e.g. inhibiteurs PCR). Afin d’améliorer les quantité et qualité d’ADN extrait, des approches de purification par billes magnétiques couplées à un automate de purification peuvent être déployées.
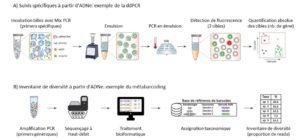
- Evolution des techniques de biologie moléculaire : le suivi des populations d’écrevisses en milieu aquatique via l’ADNe repose principalement sur deux approches de biologie moléculaire : i) le métabarcoding permettant de réaliser des inventaires de diversité en séquençage via l’utilisation de primers PCR ciblant un groupe biologique d’intérêt ; ii) l’utilisation de techniques de PCR permettant de faire des suivis semi-quantitatifs d’une espèce cible via l’utilisation de primers spécifiques (Figure 2). Ces approches sont complémentaires en termes de détection et de quantification des espèces, mais leur application opérationnelle reste encore limitée et à améliorer. En métabarcoding, de nouveaux primers ont récemment été développés pour cibler les décapodes, offrant de nouvelles perspectives de détection des espèces (Komai et al. 2019). En ce qui concerne les suivis spécifiques, l’utilisation de technique récente basée sur l’ADNe (qPCR, ddPCR) permet de mesurer plus finement la quantité d’ADN libérée par une espèce, ce qui est particulièrement adapté au suivi d’espèces peu abondante dans l’environnement comme A. pallipes ou difficilement observable comme A. astaci. De plus, cette technique peut être déployée pour suivre plusieurs espèces en parallèle, ce qui permet aussi une optimisation des coûts et temps d’analyses en laboratoire.
Objectifs et mise en place du projet
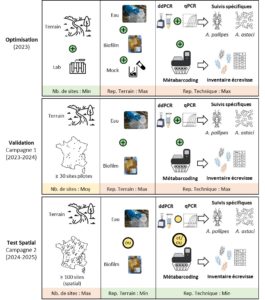
L’objectif principal de cette étude est de proposer aux gestionnaires des milieux aquatiques de nouveaux outils utilisant l’ADNe qui soient complémentaires aux approches traditionnelles et adaptés à la conservation de A. pallipes. Le projet se décompose donc en plusieurs étapes (Figure 3) :
Optimisation technique et méthodologique
- Optimiser les protocoles d’échantillonnages pour les prélèvements d’eau et de biofilm afin qu’ils puissent être mis en œuvre le plus facilement possible par les opérateurs de terrain.
- Valider les amorces et sondes ADN spécifiques à l’écrevisse à pattes blanches (A. pallipes) et à l’agent responsable de la peste de l’écrevisse (A. astaci) permettant la détection de ces espèces par qPCR et ddPCR à partir d’échantillons ADNe.
- Valider les amorces génériques permettant de réaliser des inventaires de diversité écrevisse en métabarcoding à partir d’échantillons ADNe.
- Réaliser des tests en conditions contrôlées (communautés artificielles « mock ») et sur un panel d’échantillons environnementaux (eau et biofilms en cours d’eau).
Validation de l’approche ADNe la plus opérationnelle
- Déployer toutes les approches ADNe (qPCR/ddPCR/metabarcoding) sur un panel de sites pilotes (≥30) répartis sur le territoire et présentant des contextes variés (e.g. densité de populations, hydromorphologie, géologie).
- Comparer les capacités de détection et de quantification des 2 espèces à partir de l’ADNe obtenu via des échantillons d’eau et de biofilm aquatique collectés sur différents cours d’eau.
- Si disponible, confronter les données obtenues en qPCR, ddPCR et métabarcoding sur les suivis ADNe aux données historiques de captures et de piégeages.
- Sélectionner à partir des résultats obtenus lors de la campagne 1, l’approche ADNe la plus opérationnelle (technique moléculaire, type d’échantillon, nb. de réplicats)
Test opérationnel à large échelle spatiale
- Déployer l’approche ADNe la plus opérationnelle sur un panel de sites (≥100) répartis sur le territoire et présentant des contextes variés. En fonction du nombre de sites d’étude, des approches spatiales seront privilégiées afin d’évaluer la représentativité spatiale du signal ADNe.
- Evaluer les avantages ainsi que les limites techniques et méthodologiques de l’approche ADN et ainsi statuer sur son potentiel d’application en routine.
- En fonction des attentes et besoins, possibilité de déployer l’approche sur un plus grand nombre de stations et de bassins versants à différentes périodes de l’années pour permettre un suivi de la dynamique temporelle du signal ADNe de A. pallipes et de la peste A. astaci.
Les informations obtenues en termes de détection et potentiellement de quantification des espèces (écrevisse et peste) pourront être coordonnées avec des suivis traditionnels pour affiner l’interprétation du signal ADNe. Une partie de ces suivis pourra être réalisés par les partenaires, mais les gestionnaires locaux seront sollicités pour réaliser la majorité des suivis (fédérations de pêche, parcs naturels régionaux…).
Les campagnes de prélèvements ADNe (biofilm, eau) seront principalement réalisées par les gestionnaires locaux après un accompagnement sur site (si possible) et une préparation à l’échantillonnage par visioconférence.
Les résultats obtenus à l’issue de chaque phase seront analysés au fur et à mesure de l’avancée du projet, ces derniers conditionneront le déroulement des phases suivantes. Des ajustements méthodologiques et stratégiques pourront alors être réalisés en cours de projet pour s’ajuster à la réalité des données en accord avec les objectifs du projet.
En fonction des contextes locaux et des objectifs de conservation, il pourra être envisagé de mettre en place des approches ADNe pour le suivi spécifique d’autres espèces d’écrevisses autochtones et allochtones.
Calendrier prévisionnel et résultats attendus

Table 2 – Calendrier prévisionnel présentant par trimestre les actions attendues lors des différentes phases du projet (début Janvier 2023 à fin Juin 2025).
La durée du projet porte sur une durée de 30 mois pouvant être planifié de début janvier 2023 à fin juin 2025 (Table 1). Ce calendrier reste prévisionnel et pourra évoluer en fonction des contraintes administratives et de mise en place technique.
Les premiers résultats de l’étude sont attendus pour 1er semestre 2024.
Références SCIMABIO Interface
Baudry T., Laffitte M., Noizat C., Delaunay C., Ricou G., Vasselon V. & Grandjean F. (2023). Influence of distance from source population and seasonality in eDNA detection of white‐clawed crayfish, through qPCR and ddPCR assays. Environmental DNA.
Références bibliographiques
Baudry T., Mauvisseau Q., Goût J.-P., Arqué A., Delaunay, C. Smith-Ravin, J. Sweet, M. & Grandjean F. (2021). Mapping a super-invader in a biodiversity hotspot, an eDNA-based success story. Ecological Indicators.
Chucholl F., Fiolka F., Segelbacher G. & Epp L. S. (2021). eDNA Detection of Native and Invasive Crayfish Species Allows for Year-Round Monitoring and Large-Scale Screening of Lotic Systems. Frontiers in Environmental Science.
Collas M., Burgun V., Grandjean F., Poulet N. & Penil C. (2015). La situation des écrevisses en France – Résultats de l’enquête nationale 2014. Onema.
Demaree B., Weisgerber D., Dolatmoradi A., Hatori M. & Abate A.R. (2018). Direct quantification of EGFR variant allele frequency in cell-free DNA using a microfluidic-free digital droplet PCR assay. Methods in Cell Biology.
Dunn N., Priestley V., Herraiz A., Arnold R. & Savolainen V. (2017). Behavior and season affect crayfish detection and density inference using environmental DNA. Ecology and Evolution.
Füreder L., Gherardi F., Holdich D., Reynolds J., Sibley P. & Souty-Grosset C. (2010). Austropotamobius pallipes. The IUCN Red List of Threatened Species 2010: e.T2430A9438817.
Jussila J., Edsman L., Maguire I., Diéguez-Uribeondo J. & Theissinger K. (2021). Money Kills Native Ecosystems: European Crayfish as an Example. Frontiers in Ecology and Evolution.
King AC., Krieg R., Weston A., Zenker A.K. (2022). Using eDNA to simultaneously detect the distribution of native and invasive crayfish within an entire country. Journal of Environmental Management.
Komai T., Gotoh R.O., Sado T. & Miya M. (2019). Development of a new set of PCR primers for eDNA metabarcoding decapod crustaceans. Metabarcoding and Metagenomics.
Manenti R., Barzaghi B., Nessi A., Cioccarelli S., Villa M. & Ficetola G. F. (2021). Not Only Environmental Conditions but Also Human Awareness Matters: A Successful Post-Crayfish Plague Reintroduction of the White-Clawed Crayfish (Austropotamobius pallipes) in Northern Italy. Frontiers in Ecology and Evolution.
Martín-Torrijos L., Correa-Villalona A. J., Pradillo A. & Diéguez-Uribeondo J. (2021). Coexistence of Two Invasive Species, Procambarus clarkii and Aphanomyces astaci, in Brackish Waters of a Mediterranean Coastal Lagoon. Frontiers in Ecology and Evolution.
MNHN & OFB [Ed]. 2003-2021. Fiche de Austropotamobius pallipes (Lereboullet, 1858). Inventaire national du patrimoine naturel (INPN).
Rivera S. F., Vasselon V., Mary N., Monnier O., Rimet F. & Bouchez A. (2021). Exploring the capacity of aquatic biofilms to act as environmental DNA samplers: Test on macroinvertebrate communities in rivers. Science of The Total Environment.
Rusch J. C., Mojžišová M., Strand D. A., Svobodová J., Vrålstad T. & Petrusek A. (2020). Simultaneous detection of native and invasive crayfish and Aphanomyces astaci from environmental DNA samples in a wide range of habitats in Central Europe. NeoBiota.
Troth C. R., Burian A., Mauvisseau Q., Bulling M., Nightingale J., Mauvisseau C. & Sweet M. J. (2020). Development and application of eDNA-based tools for the conservation of white-clawed crayfish. Science of The Total Environment.
Vasselon V., Rimet F., Domaizon I., Monnier O., Reyjol Y. & Bouchez A. (2019). Assessing pollution of aquatic environments with diatoms’ DNA metabarcoding: experience and developments from France water framework directive networks. Metabarcoding and Metagenomics.